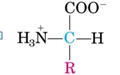
Los aminoácidos son compuestos orgánicos que se combinan
para formar proteínas. Los aminoácidos y las proteínas son los pilares
fundamentales de la vida. Es una molécula orgánica con un grupo amino (-NH2) y un grupo carboxilo (-COOH). Se pueden dividir en 20 aminoácidos.
En la siguiente tabla se presentan el nombre de los 20 aminoácidos, con sus abreviaturas de tres y una letra:
Estructura de los 20 aminoácidos:
Clasificación de los
aminoácidos:
Los aminoácidos se clasifican en: Alifáticos, Polares sin carga (neutros), Átomos de Azufre, Aromáticos,
Básicos (carga positiva), Básicos (carga negativa).
Los aminoácidos según su clasificación se pueden agrupar de
la siguiente manera:
Ionización de los
aminoácidos:
Las
propiedades físicas de los aminoácidos reciben influencias de los estados
iónicos de los grupos a-carboxilo y a-amino y de todos los grupos
ionizables que haya en las cadenas laterales. Cada grupo ionizable guarda
relación con un valor específico de pKa, que corresponde al pH al que
son iguales las concentraciones de las formas protonada y no protonada. Cuando
el pH de la solución es menor que el pKa, predomina la forma protonada y
el aminoácido es entonces un ácido real, capaz de donar un protón. Cuando el pH
de la solución es mayor que el pKa del grupo ionizable, la forma no
protonada de ese grupo predomina, y el aminoácido existe en forma de base
conjugada, que es aceptora de protones. Cada aminoácido tiene al menos dos
valores de pKa que corresponden a la ionización de los grupos
a-carboxilo y a-amino. Los estados iónicos de las cadenas laterales de los
aminoácidos influyen sobre las estructuras tridimensionales de las proteínas.
|
|
Valores del pKa
|
|
|
|
Aminoácido
|
Grupo
amino
|
Grupo ácido
|
Cadena
lateral
|
|
Alanina
|
9.9
|
2.4
|
|
|
Valina
|
9.7
|
2.3
|
|
|
Leucina
|
9.7
|
2.3
|
|
|
Isoleucina
|
9.8
|
2.3
|
|
|
Prolina
|
10.6
|
2.0
|
|
|
Triptófano
|
9.4
|
2.5
|
|
|
Fenilalanina
|
9.3
|
2.2
|
|
|
Metionina
|
9.3
|
2.1
|
|
|
Glicina
|
9.8
|
2.4
|
|
|
Serina
|
9.2
|
2.2
|
|
|
Treonina
|
9.1
|
2.1
|
|
|
Cisteína
|
10.7
|
1.9
|
8.4
|
|
Tirosina
|
9.2
|
2.2
|
10.5
|
|
Asparagina
|
8.7
|
2.1
|
|
|
Glutamina
|
9.1
|
2.2
|
|
|
Acido
Aspártico
|
9.9
|
2.0
|
3.9
|
|
Acido
Glutámico
|
9.5
|
2.1
|
4.1
|
|
Lisina
|
9.1
|
2.2
|
10.5
|
|
Histidina
|
9.3
|
1.8
|
12.5
|
|
Arginina
|
9.0
|
1.8
|
6.0
|
Los valores de pKa de los aminoácidos se determinan
con curvas de titulación. El pKa de un grupo ionizable corresponde a un
punto medio en su curva de titulación. Es el pH al cual la concentración de la
forma ácida (donador de protones) es exactamente igual a la concentración de su
base conjugada (aceptor de protones).
La característica más llamativa de los aminoácidos (AA) es la existencia en una misma molécula de
grupos ácidos (capaces de ceder H+) y grupos básicos (capaces de captar H+).
Por lo tanto, en medio ácido se comportan como bases, y en medio básico se
comportan como ácidos. Las moléculas que presentan esta característica se dice
que son anfóteros o anfolitos.
Los grupos ácidos y básicos pueden neutralizarse mutuamente,
constituyendo una sal interna formada por un ión híbrido (carga positiva y
carga negativa), que se llama zwitterión.
Si consideramos un AA sencillo, éste puede adoptar tres
formas iónicas diferentes:
El primer grupo que se disocia es el carboxilo (pK1 =
2,22). Por la proximidad del grupo NH3+, el COOH se comporta como un ácido
moderadamente fuerte. Si aplicamos la ecuación de Henderson-Hasselbalch al
primer equilibrio de disociación, resulta que a pH fisiológico (7,4), la
concentración de la forma catiónica es prácticamente despreciable (una de cada
151.000 moléculas en la forma zwitterión). El segundo grupo en disociarse es el
NH3+. Como el pK2 es 9,86, la concentración de la forma aniónica es muy pequeña
en comparación con la forma zwitterión (1 molécula en forma aniónica por cada
300 en forma zwitteriónica). El AA se comporta a pH fisiológico como un ácido
débil que está disociado al 0,35%.
Existe un pH para el cual la carga eléctrica media de
las moléculas es cero. Este pH se llama punto isoeléctrico (pI). El
pI es el pH en el que la molécula se disocia por igual en ambos sentidos, y
como equidista de los dos valores de pK, puede obtenerse por su semisuma:
 Unión de aminoácidos por enlaces peptídicos en las proteínas:
Unión de aminoácidos por enlaces peptídicos en las proteínas: La secuencia lineal de aminoácidos en una cadena polipeptídica se llama estructura primaria de una proteína. A los niveles más altos de estructura se les llaman estructura secundaria: arreglo espacial local de los átomos de la cadena del polipéptido (sin tomar en cuenta la cadena lateral), terciaria: estructura tridimensional de todo el polipéptido y cuaternaria: arreglo espacial de las subunidades de proteínas compuestas por múltiples polipéptidos.
El enlace que se forma entre los
aminoácidos es un enlace de amida y se llama enlace peptídico, o enlace
de péptido. Esta unión se puede concebir como el resultado de una
condensación simple del grupo carboxilo a de un aminoácido con el grupo amino a
del otro. A diferencia de los grupos carboxilo y amino de los aminoácidos
libres en solución, los grupos que intervienen en los enlaces peptídicos no
tienen cargas iónicas.
Las mitades de aminoácido unidas en una cadena polipeptídica se llaman residuos de aminoácido. Los nombres de los residuos se forman sustituyendo la terminación –ina o -ato por -ilo (o -il, en nombres compuestos). La terminación ilo indica que el residuo es una unidad de acilo (estructura que carece del hidroxilo del grupo carboxilo).
Las mitades de aminoácido unidas en una cadena polipeptídica se llaman residuos de aminoácido. Los nombres de los residuos se forman sustituyendo la terminación –ina o -ato por -ilo (o -il, en nombres compuestos). La terminación ilo indica que el residuo es una unidad de acilo (estructura que carece del hidroxilo del grupo carboxilo).
Técnicas de purificación de las proteínas:
Es
una serie de procesos que permiten aislar un sólo tipo de proteína de una mezcla compleja. Se pueden aplicar
pocas técnicas analíticas en forma directa a las mezclas crudas de proteínas
celulares porque contienen cientos (o miles) de proteínas diferentes. Los pasos
de purificación son distintos para cada proteína. Se determinan probando con varias
técnicas distintas hasta que se desarrolla un procedimiento que produce en
forma repetida proteína altamente purificada y que conserva su actividad
biológica. Los pasos de purificación suelen aprovechar pequeñas diferencias en
las solubilidades, cargas netas, tamaños y especificidades de unión de las
proteínas.
La mayor parte de las técnicas de purificación se lleva a cabo entre 0 y 4°C
para minimizar los procesos dependientes de la temperatura, como la degradación
y la desnaturalización (desdoblado) de la proteína. El primer paso en la
purificación de una proteína es preparar una solución de proteínas. El
siguiente paso de la purificación es, con frecuencia, una relativamente
separación cruda, o fraccionamiento, procedimiento que aprovecha las
distintas solubilidades de las proteínas en soluciones salinas. Ser reemplazados por
los solutos de la solución amortiguadora. A continuación se puede usar la cromatografía
en columna para fraccionar la mezcla de proteínas que resta después de la
precipitación con sulfato de amonio y la diálisis. Una columna cilíndrica se
llena con un material insoluble, como fibras de celulosa sustituida o
esferillas de material sintético. La mezcla de proteínas se agrega a la columna
y se lava haciendo pasar por la matriz de material insoluble un solvente. A
medida que el solvente fluye por la columna, el eluido (que es el
líquido que sale por el fondo de la columna) se recolecta en muchas fracciones.
Las técnicas cromatográficas se clasifican de acuerdo con el tipo de matriz. En
la cromatografía de intercambio iónico la matriz tiene cargas positivas
(resinas de intercambio de aniones) o cargas negativas (resinas de intercambio
de cationes). La cromatografía por filtración en gel separa a las
proteínas con base en su tamaño molecular. El gel es una matriz de esferillas
porosas. La cromatografía de afinidad es el tipo de cromatografía en
columna más selectivo. Se basa en interacciones específicas de unión entre la
proteína deseada y alguna otra molécula enlazada en forma covalente a la matriz
de la columna.
Técnicas analíticas:
 Las proteínas pueden separarse y
caracterizarse por: electroforesis
es el desplazamiento de la
proteína cargada en un campo eléctrico (gel de poliacrilamida Con SDS, dodecil
sulfato sódico).
Las proteínas pueden separarse y
caracterizarse por: electroforesis
es el desplazamiento de la
proteína cargada en un campo eléctrico (gel de poliacrilamida Con SDS, dodecil
sulfato sódico).
La espectrometría de masas, como indica
el nombre, es una técnica que determina la masa de una molécula. El tipo más
sencillo de espectrómetro de masas mide el tiempo que tarda una molécula
cargada, en fase gaseosa, para trasladarse desde su punto de inyección hasta un
detector sensible. Ese tiempo depende de la carga de una molécula y de su masa,
y el resultado se maneja como relación de masa/carga.
En la espectrometría de masas por electroaspersión,
la solución de proteína se bombea a través de una aguja metálica, a alto
voltaje, para crear diminutas gotitas.







No hay comentarios.:
Publicar un comentario