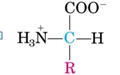
Son
un tipo de moléculas formadas por la unión de varios aminoácidos mediante enlaces
peptídicos.
Estructura
tridimensional:
Una conformación es un ordenamiento espacial de átomos
que depende de la rotación de uno o varios enlaces. La conformación de
una molécula, como la de una proteína, puede cambiar sin que los enlaces
covalentes se rompan, mientras que las diversas configuraciones de una
molécula sólo se pueden cambiar si se rompen y vuelven a unir enlaces
covalentes. La función biológica de una proteína depende por completo de su
conformación nativa.
Una proteína puede ser una sola cadena polipeptídica o puede estar formada por varias de esas cadenas unidas entre sí por interacciones débiles. El estudio de grandes conjuntos de proteínas, como el de todo el complemento de proteínas producidas por una célula, es parte de un campo emergente llamado proteómica.
Las proteínas tienen diversas formas. Muchas son macromoléculas aproximadamente esféricas, hidrosolubles y compactas cuyas cadenas polipeptídicas están dobladas de manera apretada. Esas proteínas globulares tienen un interior hidrofóbico y una superficie hidrofílica, en forma característica. Poseen penetraciones o fisuras que reconocen en forma específica a otros compuestos y se unen a ellos en forma transitoria.
Una proteína puede ser una sola cadena polipeptídica o puede estar formada por varias de esas cadenas unidas entre sí por interacciones débiles. El estudio de grandes conjuntos de proteínas, como el de todo el complemento de proteínas producidas por una célula, es parte de un campo emergente llamado proteómica.
Las proteínas tienen diversas formas. Muchas son macromoléculas aproximadamente esféricas, hidrosolubles y compactas cuyas cadenas polipeptídicas están dobladas de manera apretada. Esas proteínas globulares tienen un interior hidrofóbico y una superficie hidrofílica, en forma característica. Poseen penetraciones o fisuras que reconocen en forma específica a otros compuestos y se unen a ellos en forma transitoria.
También los polipéptidos pueden ser
partes de grandes estructuras subcelulares o extracelulares, como ribosomas,
flagelos y cilios, músculos y cromatina. Las proteínas fibrosas son
una clase particular de proteínas estructurales que proporcionan soporte
mecánico a las células u organismos. En el caso típico, las proteínas fibrosas
se ensamblan en grandes cables o hebras. Como ejemplos de proteínas fibrosas
están la a-queratina, el componente principal de cabello y uñas, y la colágena,
el componente proteínico principal de tendones, piel, huesos y dientes.
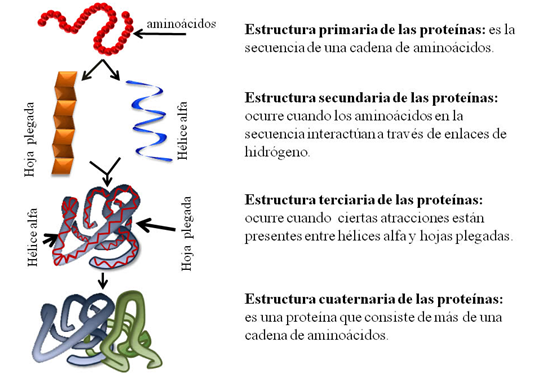
Niveles de estructura de las proteínas:
Métodos para determinar
la estructura de las proteínas:
Conformación tridimensional de una
proteína se determina por cristalografía
con rayos X.
Otra técnica para
analizar la estructura macromolecular de las proteínas es la espectroscopia de
resonancia magnética nuclear (RMN): En general, los espectros de RMN de pequeñas proteínas, como la ribonucleasa
A, se pueden deducir con facilidad, pero el espectro de una molécula grande
puede ser en extremo complicado.
Por esta razón es muy difícil determina la estructura de las proteínas mayores,
pero la técnica es muy poderosa para las proteínas más.
Por esta razón es muy difícil determina la estructura de las proteínas mayores,
pero la técnica es muy poderosa para las proteínas más.
Conformación del grupo peptídico:
La estructura de las proteínas comienza con la de los enlaces
peptídicos, o enlaces de péptido, que unen a los aminoácidos en una cadena
polipeptídica. Los dos átomos que intervienen en el enlace peptídico, junto con
sus cuatro sustituyentes (el átomo de oxígeno carbonílico, el átomo de
hidrógeno de amida y los dos átomos adyacentes de carbono a) constituyen el
grupo peptídico. Los análisis cristalográficos de pequeños péptidos con rayos X
revelan que el enlace entre el carbono carbonílico y el nitrógeno es más corto
que un enlace sencillo típico C—N, pero más
largo que los dobles enlaces CN típicos. Además, el enlace entre el
carbono carbonílico y el oxígeno es un poco mayor que el doble enlace típico
CO. Esas mediciones indican que los enlaces peptídicos tienen ciertas
propiedades del enlace doble y se pueden representar mejor como un híbrido de
resonancia. En la conformación trans, los dos
carbonos a de residuos adyacentes
de aminoácido están en lados opuestos del enlace peptídico y en las esquinas opuestas
del rectángulo que forma el grupo peptídico plano. En la conformación cis, los dos carbonos a están en el mismo lado del enlace peptídico y están más cerca entre sí. Las conformaciones cis y trans se producen durante la síntesis de la proteína, cuando el enlace peptídico se forma uniendo dos aminoácidos a la cadena polipeptídica. Las dos conformaciones no se pueden interconvertir por giro respecto al enlace peptídico, una vez formado. La conformación cis es menos favorable que la conformación trans, que es
extendida debido a impedimentos estéricos entre las cadenas laterales unidas a los dos
átomos de carbono a. En consecuencia, casi todos los grupos peptídicos en las proteí-
nas tienen la conformación trans.
de aminoácido están en lados opuestos del enlace peptídico y en las esquinas opuestas
del rectángulo que forma el grupo peptídico plano. En la conformación cis, los dos carbonos a están en el mismo lado del enlace peptídico y están más cerca entre sí. Las conformaciones cis y trans se producen durante la síntesis de la proteína, cuando el enlace peptídico se forma uniendo dos aminoácidos a la cadena polipeptídica. Las dos conformaciones no se pueden interconvertir por giro respecto al enlace peptídico, una vez formado. La conformación cis es menos favorable que la conformación trans, que es
extendida debido a impedimentos estéricos entre las cadenas laterales unidas a los dos
átomos de carbono a. En consecuencia, casi todos los grupos peptídicos en las proteí-
nas tienen la conformación trans.
La hélice a:
Una hélice a puede ser
una rosca izquierda o derecha. Las hélices a que se encuentran en las proteínas
casi siempre son derecha. Los puentes de hidrógeno entre los residuos de aminoácido
tienen estabilidad especial en el interior hidrofóbico de una proteína, donde
las moléculas de agua no entran y en consecuencia no pueden competir en la
formación de puentes. En una hélice a, todos los grupos carbonilo apuntan hacia
el C-terminal. Ya que cada grupo peptídico es polar y todos los puentes de
hidrógeno apuntan en la misma dirección, toda la hélice es un dipolo con un
N-terminal positivo y un C-terminal negativo.
Hebras b y láminas b:
La otra estructura
secundaria común se llama estructura b, una clase que incluye a hebras b y
láminas b. Las hebras B son partes de la cadena polipeptídica que se
encuentran casi totalmente extendidas. Cada residuo en una hebra b ocupa de
0.32 a 0.34 nm de la longitud total, en contraste con la espiral compacta de
una hélice a, donde cada residuo corresponde a 0.15 nm de la longitud general.
Cuando se ordenan varias hebras b lado a lado forman láminas B. Las
hebras b en una lámina pueden ser paralelas (con la misma dirección de N a
C-terminal) o antiparalelas (con direcciones opuestas de N a C-terminal).
 Cuando las hebras b son antiparalelas, los puentes de
hidrógeno son casi perpendiculares a las cadenas extendidas del polipéptido.
Nótese que en la lámina b antiparalela los átomos de oxígeno carbonílico y los
de hidrógeno de amida de un residuo forman puentes de hidrógeno con el
hidrógeno de amida y el oxígeno carbonílico de un
Cuando las hebras b son antiparalelas, los puentes de
hidrógeno son casi perpendiculares a las cadenas extendidas del polipéptido.
Nótese que en la lámina b antiparalela los átomos de oxígeno carbonílico y los
de hidrógeno de amida de un residuo forman puentes de hidrógeno con el
hidrógeno de amida y el oxígeno carbonílico de unsolo residuo en la otra hebra.
Las láminas paralelas son menos estables que
las antiparalelas, quizá porque los puentes de hidrógeno están distorsionados
en el ordenamiento paralelo. A veces, a la lámina b se le llama lámina B
plegada ya que los grupos peptídicos planos se encuentran entre sí formando
ángulos como en el plisado de un acordeón.
Asas y
giros:
Las asas y los giros
unen a hélices a y hebras b y permiten que la cadena de polipéptido se doble
sobre sí misma para producir la forma tridimensional compacta que se ve en la
estructura nativa. Las asas contienen con frecuencia residuos hidrofílicos y
se suelen encontrar en las superficies de las proteínas, donde están expuestas
al solvente y forman puentes de hidrógeno con el agua. Las asas que sólo
contienen pocos (hasta cinco) residuos se llaman giros si causan un
cambio abrupto en la dirección de una cadena de polipéptidos. Los tipos más
comunes de giros bruscos se llaman giros inversos o giros B
porque con frecuencia conectan hebras b antiparalelas diferentes. Hay dos tipos
comunes de giro b que se designan tipo I y tipo II. Ambos contienen cuatro
residuos de aminoácidos y los estabilizan puentes de hidrógeno entre el oxígeno
carbonílico del primer residuo y el hidrógeno de amida del cuarto residuo. Giro tipo I. La
estructura se halla estabilizada por un puente de hidrógeno entre el oxígeno
carbonílico del primer residuo N-terminal (Phe) y el hidrógeno de amida del
cuarto residuo (Gly). Obsérvese el residuo de prolina en la posición n + 1.
Giro tipo II. También está estabilizado por un puente de hidrógeno entre el oxígeno carbonílico del primer residuo N-terminal (Val) y el hidrógeno de amida del cuarto residuo (Asn).
Giro tipo II. También está estabilizado por un puente de hidrógeno entre el oxígeno carbonílico del primer residuo N-terminal (Val) y el hidrógeno de amida del cuarto residuo (Asn).
Estructura terciaria de las proteínas:
Describe la cadena polipetídica
totalmente plegada y compactada (estructura tridimensional del polipéptido). Una
propiedad importante de la estructura terciaria es que los residuos de
aminoácidos alejados en la estructura primaria se acercan entre sí y permiten
interacciones entre sus cadenas laterales. Mientras que la estructura
secundaria está estabilizada por puentes de hidrógeno entre los hidrógenos de
amida y oxígenos carbonílicos de la columna vertebral del polipéptido, la
estructura terciaria se halla estabilizada en especial por interacciones no
covalentes.
Estructuras supersecundarias:
Las estructuras supersecundarias, o motivos, son combinaciones reconocibles de hélices a, hebras b y giros que aparecen en diversas proteínas. A veces los motivos se relacionan con determinada función, aunque los motivos de estructura similar pueden tener funciones distintas en proteínas diferentes.
Las estructuras supersecundarias, o motivos, son combinaciones reconocibles de hélices a, hebras b y giros que aparecen en diversas proteínas. A veces los motivos se relacionan con determinada función, aunque los motivos de estructura similar pueden tener funciones distintas en proteínas diferentes.
Dominios
Hay muchas proteínas que están formadas por varias unidades compactas, discretas,
plegadas en forma independiente llamadas dominios. Los dominios pueden consistir en
combinaciones de motivos. El tamaño de un dominio varía desde unos 25 a 30 residuos
de aminoácidos hasta más de 300. Los dominios
están unidos por asas, pero también se unen entre sí mediante interacciones débiles formadas por las cadenas laterales de aminoácidos en la superficie de cada dominio.
Hay muchas proteínas que están formadas por varias unidades compactas, discretas,
plegadas en forma independiente llamadas dominios. Los dominios pueden consistir en
combinaciones de motivos. El tamaño de un dominio varía desde unos 25 a 30 residuos
de aminoácidos hasta más de 300. Los dominios
están unidos por asas, pero también se unen entre sí mediante interacciones débiles formadas por las cadenas laterales de aminoácidos en la superficie de cada dominio.
Estructura y función de los dominios:
Es compleja la relación entre estructura y función de un dominio. Con frecuencia, un solo dominio tiene determinada función, como por ejemplo unirse a pequeñas moléculas o catalizar una sola reacción. En las enzimas multifuncionales, cada actividad catalítica puede estar asociada con uno de varios dominios presentes en una sola cadena polipeptídica. Las formas únicas de las proteínas, con sus penetraciones, interfases entre dominios y otras grietas les permiten efectuar funciones dinámicas al unirse con otras moléculas en forma selectiva y transitoria.
Es compleja la relación entre estructura y función de un dominio. Con frecuencia, un solo dominio tiene determinada función, como por ejemplo unirse a pequeñas moléculas o catalizar una sola reacción. En las enzimas multifuncionales, cada actividad catalítica puede estar asociada con uno de varios dominios presentes en una sola cadena polipeptídica. Las formas únicas de las proteínas, con sus penetraciones, interfases entre dominios y otras grietas les permiten efectuar funciones dinámicas al unirse con otras moléculas en forma selectiva y transitoria.
Estructura cuaternaria:
Se refiere a la organización y el
reordenamiento de las subunidades en una proteína con múltiples sub-unidades. Cada
subunidad es una cadena polipeptídica aparte. Los cambios en el ambiente o los tratamientos
químicos pueden alterar la conformación nativa de una proteína con la pérdida
concomitante de su actividad biológica. Esa alteración se llama desnaturalización.
La cantidad de energía necesaria para causar la desnaturalización es pequeña,
con frecuencia, quizá la equivalente a la que se necesita para alterar tres o
cuatro puentes de hidrógeno.